Ludzie cierpią na różne choroby, z których tylko część można wyleczyć istniejącymi obecnie lekami. Niestety, niektóre leki, które są już stosowane w leczeniu poszczególnych chorób, mogą być nieskuteczne i mają nieprzyjemne skutki uboczne. Nie jest zatem zaskoczeniem, że odkrywanie nowych leków jest niezwykle dynamiczną dziedziną badań, zarówno dla środowiska akademickiego, jak i przemysłu farmaceutycznego.
Opracowywanie nowych lekarstw jest złożonym i wielopłaszczyznowym problemem. Wprowadzenie nowego leku na rynek, które obejmuje badania, eksperymenty i testy, może zająć ponad dziesięć lat. W tym procesie coraz większą rolę odgrywają (super)komputery. Okazało się, że dzięki nim można znacznie zmniejszyć liczbę potencjalnych “kandydatów na leki”, oszczędzając w ten sposób ogromną ilość czasu i kosztów związanych z syntezą i testowaniem metodą prób i błędów. Mówimy o “zawężaniu” zamiast identyfikowaniu najlepszego “kandydata”, ponieważ same komputery (w najbliższej przyszłości) nie są w stanie uchwycić pełnej złożoności interakcji lekowych w organizmie człowieka. “Kandydat” musi być nie tylko skuteczny w leczeniu choroby docelowej, ale także powinien powodować jak najmniej skutków ubocznych, być stabilny chemicznie i metabolicznie, nietoksyczny (dotyczy to również jego metabolitów) oraz musi mieć wiele innych korzystnych właściwości, takich jak rozpuszczalność, zdolność do przenikania przez błony komórkowe, itp.
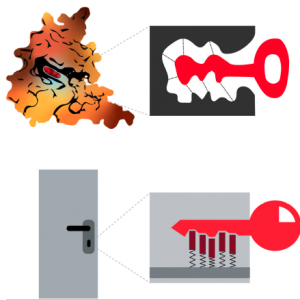
Typowy proces projektowania leku ukierunkowanego na konkretną chorobę przebiega w następujący sposób: najpierw należy zidentyfikować tzw. cel biologiczny. Celem biologicznym jest zazwyczaj receptor białkowy, enzym lub kwas nukleinowy, który odgrywa kluczową rolę w chorobie lub przetrwaniu patogenu w organizmie. Lek, często nazywany ligandem, jest zazwyczaj małą cząsteczką organiczną, która jest w stanie związać się z celem biologicznym (poprzez tzw. model klucza i zamka). Poprzez wiązanie liganda (odwracalne lub nieodwracalne), funkcja celu biologicznego ulega zmianie. Może to obejmować hamowanie enzymu poprzez wiązanie się z aktywnym miejscem, blokowanie lub aktywowanie receptorów białkowych lub oddziaływanie na kanał jonowy.
Główną rolą komputerów w odkrywaniu nowych leków jest ilościowe opisanie wiązania ligandów z określonymi celami biologicznymi. Stosuje się szerokie spektrum metod obliczeniowych, różniących się zakresem doświadczalności (tj. nie mających solidnego zaplecza teoretycznego w dziedzinie fizyki lub chemii) oraz wydajnością. Dokładność danej metody oraz jej wydajność są, niestety, odwrotnie proporcjonalne.
Szybkie, ale empiryczne metody przesiewowe są wykorzystywane do sprawdzania ogromnych baz danych znanych związków chemicznych lub cząsteczek, które można z tych związków z łatwością syntetyzować. Metody te oparte są albo na formułach empirycznych, albo na bardziej zaawansowanych metodach uczenia maszynowego/sieci neuronowych realizowanych w oparciu o istniejące dane. Z drugiej strony, najbardziej wyrafinowane podejścia naukowe opierają się na solidnych podstawach teoretycznych: chemii kwantowej i termodynamice statystycznej. Testowane są zatem aktualne możliwości superkomputerów, ale gdy maszyny te będą już wystarczająco wydajne, rozpocznie się zupełnie nowa era projektowania leków.

